Real-Time PCR预混液(染料法,含指示剂)是采用SYBR Green I嵌合荧光法进行Real-Time PCR的专用试剂。产品中预先混有Real-Time PCR反应所需合适浓度的SYBR Green I,是一种2×浓度的qPCR预混液。本制品额外添加了指示剂,利于大量样品的加样,减少误操作概率。
2×SuperReal SYBR Color qPCR Mix采用了独特的双组分热启动DNA聚合酶(化学修饰的HotStar Taq DNA聚合酶和抗体修饰的Anti Taq DNA聚合酶),配合精心优化buffer体系,具有高扩增效率,高扩增特异性和宽广的可信范围的特点。本升级版试剂在经过成分调整后,扩增特异性上的优势更为突出。

- 2×SuperReal SYBR Color qPCR Mix采用了独特的双组分热启动DNA聚合酶(化学修饰的HotStar Taq DNA聚合酶和抗体修饰的Anti Taq DNA聚合酶),从而构成酶活自动调节系统,配合精心优化buffer体系,具有高扩增效率,高扩增特异性和宽广的可信范围的特点。
- 本制品Buffer体系平衡了K+和NH4+的比例,还特别添加了独特的H-Bond因子,能协同调整反应体系中的氢键作用力,使引物模板退火条件更加严谨,反应的专一性增强,重复性更好。
- 2×SuperReal SYBR Color qPCR Mix中预混有SYBR Green I,PCR反应液配制时,只需加入模板、引物、无RNA酶超纯水便可进行Real-Time PCR反应,操作简单方便。
- 本制品附带ROX Reference Dye,用于消除信号本底以及校正孔与孔之间产生的荧光信号误差,方便客户针对不同型号荧光定量PCR仪时选择对应浓度使用。
- 添加颜色指示剂,令加样更加简便,有效降低误操作概率。经过严格检测,指示剂不会影响染料的荧光值和定量结果。


本制品采用了独特的双组分热启动DNA聚合酶进行PCR扩增,通过检测反应进程中SYBR Green I的荧光强度,达到检测PCR产物扩增量的目的。
本制品中双热启动酶构成了独特的酶活自动调节系统。酶活自动调节系统是由化学修饰的HotStar Taq DNA聚合酶和抗体修饰的Anti Taq DNA聚合酶组成。其中HotStar Taq DNA聚合酶占绝大部分比例,其聚合酶活性的激活是严格依赖于95℃高温的一个缓释过程,而Anti Taq DNA聚合酶则在95℃高温下完全激活。经过95℃条件下孵育15min激活大部分HotStarTaq DNA聚合酶,进入PCR循环后,每经过一轮95℃条件下变性,即可重新激活一部分HotStar Taq DNA聚合酶。HotStar Taq DNA聚合酶独特的酶活缓释机制使其可以与Anti Taq DNA聚合酶构成独特的酶活自动调节系统。PCR反应初期,完全激活的Anti Taq DNA聚合酶可以协同已经激活的HotStar Taq DNA聚合酶达到优化的酶活状态,而在整个PCR反应过程中,每一轮新释放的HotStart Taq DNA聚合酶活力刚好可以弥补因热变性所导致的部分酶活损失。因此,SuperReal SYBR Color qPCR Mix在整个PCR反应过程始终保持优良的DNA聚合酶活力,配合精心优化Buffer体系,从而可以获得高扩增效率,高扩增特异性和更加广泛性的模板适应性。

| 组分 | 500T | 5000T |
| 2×SuperReal SYBR Color qPCR Mix | 4×1.25mL | 40×1.25mL |
| 50×ROX Reference Dye | 1mL | 10×1mL |
| 40×Dilution Buffer | 1.25mL | 10×1.25mL |
| RNase-Free ddH2O | 5×1mL | 50×1mL |
保存:-20℃,有效期1年。

收到本制品后,请立即置于-30~-15℃避光保存。从-30~-15℃取出使用时,将冻存的2×SuperReal SYBR Color qPCR Mix和50×ROX Reference Dye融解,然后轻轻颠倒混匀,待溶液完全均一后再行使用。如解冻后没有使用,须彻底混匀后重新冷冻(在解冻过程中盐会出现分层现象,未混匀进行冷冻,盐晶体的析出将会对酶造成损害)。如需一段时间内经常取用,可在2~8℃条件下储存3个月。避免反复多次冻融。

- PCR反应液中各颜色指示剂的终浓度应为1×,请根据模板的添加量计算Dilution Buffer的用量。
- PCR反应的预变性条件必须设定为95℃ 15min,用以充分激活热启动酶。
- 本制品中含有荧光染料SYBR Green I,保存本制品或配制PCR反应液时应避免强光照射。
- 如果试剂没有混匀,其反应性能会有所下降。使用时请上下颠倒轻轻混匀,请不要使用涡旋仪进行混匀,尽量避免出现泡沫,并经瞬时离心后使用。
- 引物终浓度为0.3μM可以在大多数体系中获得良好的扩增结果。如果需要进一步优化,可以在0.2~0.5μM范围内调整引物浓度。
- 20μL反应体系中,基因组DNA或cDNA模板的使用量一般小于100ng,逆转录产物作为模板时,使用量应不超过PCR体系终体积的20%。

- 建立Real-Time PCR反应体系
- 解冻2×SuperReal SYBR Color qPCR Mix、50×ROX Reference Dye、模板、引物、40×Dilution Buffer和RNase-Free ddH2O,并将所有试剂在室温下平衡并彻底混匀。
- 建议置于冰上进行Real-Time PCR反应液的配制。
40×Dilution Buffer为黄色,2×SuperReal SYBR Color qPCR Mix为蓝色。如需稀释模板,则最终反应体系应为绿色;否则应为蓝色。如颜色不符,请检查体系中是否正确加入相关组分。成分 50μL体系 25μL体系 20μL体系 终浓度 2×SuperReal SYBR Color qPCR Mix 25μL 12.5μL 10μL 1× 正向引物(10μM) 1.5μL 0.75μL 0.6μL 0.3μM 反向引物(10μM) 1.5μL 0.75μL 0.6μL 0.3μM cDNA模板(含Dilution Buffer) - - - -ng-pg 50×ROX Reference Dye - - - - RNase-free ddH2O 至50μL 至25μL 至20μL - - 引物终浓度为0.3μM可以在大多数体系中获得良好的扩增结果。扩增效率不高时,可增加PCR反应体系中的引物浓度;发生非特异扩增时,可适当减少PCR反应体系中的引物浓度。需要进一步优化引物浓度的,可以在0.2~0.5μM范围内调整。
- 如需稀释模板,应先将cDNA模板与40×Dilution Buffer按一定比例混合成稀释模板。稀释比例如下:
- 20μL反应体系中cDNA用量与40×Dilution Buffer含量对应表:
20μL PCR体系中稀释
模板的添加量(μL)1 2 2.5 3 4 5 6 稀释模板中Dilution
Buffer的浓度20× 10× 8× 6.7× 5× 4× 3.3× 100μL稀释模板中所需40×
Dilution Buffer的量(μL)50 25 20 16.7 12.5 10 8.4 100μL稀释模板中所需
cDNA的量(μL)50 75 80 83.3 87.5 90 91.6 - 50μL反应体系中cDNA用量与40×Dilution Buffer含量对应表:
50μL PCR体系中稀释
模板的添加量(μL)2 2.5 3 4 5 6 8 稀释模板中Dilution
Buffer的浓度25× 20× 16.7× 12.5× 10× 8.3× 6.25× 100μL稀释模板中所需40×
Dilution Buffer的量(μL)62.5 50 41.7 31.3 25 20.8 15.6 100μL稀释模板中所需
cDNA的量(μL)37.5 50 58.3 68.7 75 79.2 84.4
- 20μL反应体系中cDNA用量与40×Dilution Buffer含量对应表:
- 几种常见仪器的匹配ROX Reference Dye浓度见下表:
仪器 终浓度 ABI PRISM 7000/7300/7700/7900HT/StepOne等 5×(例如5μL ROX/50μL体系) ABI 7500、7500 Fast;Stratagene Mx3000P、Mx3005P和Mx4000等 1×(例如1μL ROX/50μL体系) Roche仪器,Bio-Rad仪器,Eppendorf仪器等 无需添加
- 引物终浓度为0.3μM可以在大多数体系中获得良好的扩增结果。扩增效率不高时,可增加PCR反应体系中的引物浓度;发生非特异扩增时,可适当减少PCR反应体系中的引物浓度。需要进一步优化引物浓度的,可以在0.2~0.5μM范围内调整。
- 解冻2×SuperReal SYBR Color qPCR Mix、50×ROX Reference Dye、模板、引物、40×Dilution Buffer和RNase-Free ddH2O,并将所有试剂在室温下平衡并彻底混匀。
- 进行Real-Time PCR反应
- 建议采用两步法PCR反应程序进行反应。当出现模板浓度过低引起非特异扩增,引物Tm值较低导致的扩增效率低下或扩增曲线重现性不佳等现象时,建议尝试进行三步法PCR扩增反应。
- 两步法反应程序:
阶段 循环 温度 时间 内容 荧光信号采集 预变性 1 95℃ 15min 预变性 否 PCR反应 40 95℃ 10sec 变性 否 60~66℃▲1 20~32sec▲3 退火/延伸 是 熔解曲线分析(Melting/Dissociation Curve Stage) - 三步法反应程序:
阶段 循环 温度 时间 内容 荧光信号采集 预变性 1 95℃ 15min 预变性 否 PCR反应 40 95℃ 10sec 变性 否 50~60℃▲2 20sec 退火 否 72℃ 20~32sec▲3 延伸 是 熔解曲线分析(Melting/Dissociation Curve Stage)
注▲2:通常引物退火温度比引物的解链温度(Tm)低5℃,如果引物碱基数较少,可以适当提高退火温度,这样可以使PCR的特异性增加;如果碱基数较多,那么可以适当减低退火温度。
注▲3:使用不同仪器进行时间设定时,请按照仪器使用说明书要求进行实验操作,几种常见仪器的时间设定见下表:使用时Roche LightCycler/LightCycler 480请设定在20sec。 使用ABI 7500 Fast/7900HT/7900HT Fast/ViiA 7/StepOne/StepOnePlus时请设定在30sec。 使用ABI 7000和7300时请设定在31sec。 使用ABI 7500时请设定在32sec。 - 两步法反应程序:
- 盖上反应管,轻柔混匀。可短暂离心,确保所有组分均在管底。
- 将反应体系置于荧光定量PCR仪中,开始反应。
- 以使用ABI 7500 Real-Time PCR扩增仪为例,进行扩增效率优化时,可以按照下表中所示优化方案1或优化方案2的反应条件进行反应:
基本反应程序 优化方案1
(增加两步法延伸时间进行优化)优化方案2
(使用三步法进行PCR反应)1 95℃ 15min 15min 95℃ 15min 40 95℃ 10sec 10sec 95℃ 10sec 60℃ 32sec 32~60sec 55℃ 30sec NA 72℃ 32sec - 以使用ABI 7500 Real-Time PCR扩增仪为例,进行特异性优化的参考方案:
基本反应程序 特异性优化方案
(提高两步法退火温度)循环 温度 时间 温度 时间 1 95℃ 15min 95℃ 15min 40 95℃ 10sec 95℃ 10sec 60℃ 32sec 60~64℃ 32sec
- 以使用ABI 7500 Real-Time PCR扩增仪为例,进行扩增效率优化时,可以按照下表中所示优化方案1或优化方案2的反应条件进行反应:
- 建议采用两步法PCR反应程序进行反应。当出现模板浓度过低引起非特异扩增,引物Tm值较低导致的扩增效率低下或扩增曲线重现性不佳等现象时,建议尝试进行三步法PCR扩增反应。

进行RT-qPCR反应时的操作建议:
进行RT-PCR反应时,有三种cDNA第一链合成试剂盒可以选择,分别是反转录试剂盒,cDNA第一链合成反应预混液(去除基因组DNA)和cDNA第一链合成试剂盒。其中Quicking cDNA第一链合成试剂盒是专为两步法RT-PCR第一步实验配制的,具有高灵敏度的RT-PCR反应系统,可以从微量的总RNA或poly (A)+ RNA合成第一链cDNA。该试剂盒中使用的逆转录酶King Reverse Transcriptase与通常使用的Moloney鼠白血病病毒来源的M-MLV和鸟成髓细胞病毒来源的AMV不同,是一种使用大肠杆菌工程菌进行重组表达的全新高效逆转录酶。该酶能够高效反转录多种RNA模板,高效地将RNA反转录成cDNA第一链。
以反转录试剂盒为例,下列操作步骤适用于模板量为50ng~2μg的总RNA。
- 将模板RNA在冰上解冻;5×gDNA Buffer、FQ-RT Primer Mix、10×King RT Buffer、RNase-Free ddH2O在室温(15~30℃)解冻,解冻后迅速置于冰上。使用前将每种溶液涡旋振荡混匀,简短离心以收集残留在管壁的液体。
- 以下操作步骤请在冰上进行。为了保证反应液配制的准确性,进行各项反应时,应先配制成Mix,然后再分装到每个反应管中。
- 按照表1的基因组DNA的去除体系配制混合液,彻底混匀。简短离心,并置于42℃,孵育3min。然后置于冰上放置。
表1:gDNA去除反应体系成分 用量 5×gDNA Buffer 2μL Total RNA - RNase-Free ddH2O 至10μL - 按照表2的反转录反应体系配制混合液。
表2:反转录反应体系成分 用量 10×King RT Buffer 2μL Quicking RT Enzyme Mix 1μL FQ-RT Primer Mix 2μL RNase-Free ddH2O 至10μL - 将反转录反应中的Mix加到gDNA去除步骤的反应液中,充分混匀。
- 42℃,孵育15min;95℃,孵育3min。
- 进行PCR反应
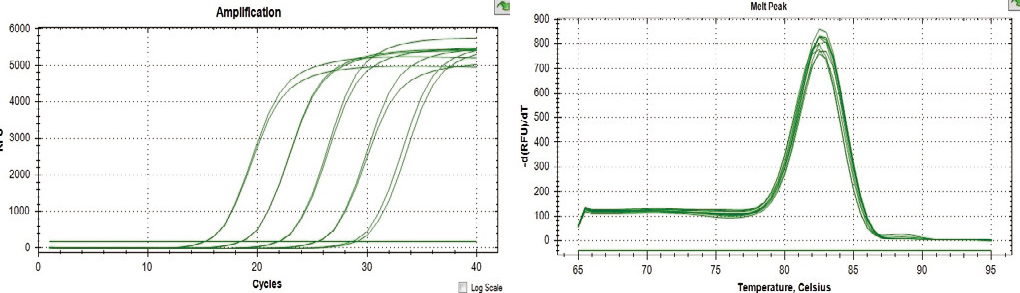
本实验是应用两步法定量RT-PCR对人Jurkat cell的Hsc mRNA进行定量检测。荧光定量PCR是使用2×SuperReal SYBR Color qPCR Mix。cDNA合成是使用反转录试剂盒进行,cDNA用量相当于100ng~10pg总RNA。扩增曲线(左图)和熔解曲线(右图)显示扩增效率和特异性良好。

进行Real-Time PCR反应时,PCR引物的设计非常重要。设计PCR扩增效率高,反应特异性强的引物可以参考以下要求。
| 引物长度 | 18~30个碱基 |
| GC含量 | 40~60% |
| Tm值 | 引物软件都可以给出Tm,与引物长度,碱基组成,引物使用缓冲的离子强度也有关。 上下游引物的Tm值要尽量接近。 简单的Tm计算公式为:Tm = 4℃(G + C)+ 2℃(A + T)。 一般采用较引物Tm值低5℃作为PCR退火温度。 提高退火温度可以增加PCR反应的特异性。 |
| 引物及PCR扩增 产物序列 | PCR扩增产物适宜长度在80~200bp之间。 尽量避开在模板的二级结构区域设计引物。 避免上下游引物3'端之间形成2个或以上的互补碱基以减少引物二聚体的形成。 引物3'端碱基不能有多于3个连续的G或C。 引物自身不应存在互补序列,否则引物自身会折叠成发夹状结构。 避免引物3'末端碱基为T。 引物序列中A、T、G、C要尽量均匀分布。 |

- 无扩增信号或扩增曲线起峰晚或仅有引物二聚体
原因 解决办法 DNA模板中存在抑制剂 重新纯化模板或降低模板使用量 Mg2+浓度不合适 使用2×SuperReal SYBR Color qPCR Mix时,PCR反应体系中Mg2+的终浓度为2mM。对有些扩增体系,可以将Mg2+终浓度提高到5mM。进行Mg2+终浓度优化时,建议每次增加0.5mM Mg2+浓度进行实验。 加样错误或试剂问题 检查试剂浓度和保存条件,包括所使用的引物和模板。重复进行实验。 热启动酶未能激活 使用试剂时,请确保预变性条件为95℃ 15min,用以有效激活热启动DNA聚合酶。 PCR条件、引物序列或浓度不当 请确认引物未发生降解,引物浓度及PCR条件,扩增不好时,通常先尝试降低退火温度,延长退火时间和提高引物浓度,有时也可以提高退火温度,增加延伸时间,降低升温速度。对于GC含量高的模板,可以适当延长变性时间。如果还是扩增不好,请重新设计引物。 起始模板问题 检查起始模板的浓度,保存条件和质量。重新对模板进行线性梯度稀释,并用新稀释模板进行实验。增加起始模板使用量。 - NTC出现较高的荧光值
原因 解决办法 试剂污染 建议使用新试剂进行实验。 PCR反应液配制时发生污染 采取必要的防污染策略(如使用带滤芯的枪头)。 引物出现降解 可以使用变性聚丙烯酰胺胶检测引物降解情况。 - 出现引物二聚体和(或)非特异扩增
原因 解决办法 Mg2+浓度不合适 使用2×SuperReal SYBR Color qPCR Mix的反应体系含有Mg2+的终浓度为2mM。对有些扩增体系,可以将Mg2+终浓度增加到5mM。建议每次增加0.5mM Mg2+浓度进行优化。 PCR退火温度太低 建议每次增加2℃进行退火温度优化。 引物设计不合适 考虑重新设计引物序列。 PCR产物太长 荧光定量PCR产物长度最好在100~150bp之间,而且不应该超过500bp。 引物出现降解 可以使用变性聚丙烯酰胺胶检测引物降解情况。 计量误差 反应体积太小会导致检测精度下降。请根据定量PCR仪推荐的反应体积重新实验。 - 定量值重现性差
原因 解决办法 仪器方面的故障 因为仪器的不适用,在温度管理或检测时产生重现性差。请根据相应仪器的说明书进行点检。 样品纯度不好 不纯的样品会导致实验的重现性差。 稀释的模板放置太久 通过梯度稀释的模板最好现配现用。 引物质量下降 尽量避免新合成引物批次间的差异,可以使用原来质量好的引物做为对照。 PCR反应条件、引物浓度、序列等不恰当 扩增效率差的PCR较容易产生重现性差。通过变更引物的度或PCR反应条件来进行调整。扩增不好时,一般可降低退火温度或提高引物浓度,也可以延长延伸时间。如模板的GC含量较高,可延长变性时间。仍得不到改善时,建议重新设计引物。 计量误差 反应体积太小会导致检测精度下降。请根据定量PCR仪推荐的反应体积重新实验。
相关搜索:Real-Time PCR预混液(染料法,含指示剂),Real-Time PCR,qPCR预混液,qPCR Mix,qPCR预混反应液,qPCR反应预混液,染料法,嵌合荧光法,SYBR Green I,含指示剂,荧光定量PCR,2×SuperReal SYBR Color qPCR Mix



